Link: https://www.cd-genomics.com/summary-of-crispr-cas9-off-target-detection-methods.html
快速概览
01 CRISPR-Cas9 脱靶检测的测序方法
02 CRISPR-Cas9 脱靶检测的计算模拟方法
基因组编辑技术处于当前生命科学研究的前沿。然而,要成功将其转化为临床应用,准确检测脱靶效应至关重要。正确评估和减轻脱靶效应是一个紧迫的问题。CRISPR-Cas9 基因编辑技术中的脱靶效应问题一直是长期关注的焦点。检测 CRISPR-Cas9 脱靶效应的主要方法有两种:计算模拟方法和测序方法 [1]。
CRISPR-Cas9 脱靶检测的测序方法
全基因组测序 (WGS)
全基因组测序 (WGS) 在评估 Cas9 诱导的脱靶突变方面与最小的评估偏差相关联。目前,通常采用 30-60x 的测序深度。然而,WGS 成本较高,较低的测序深度可能仅测序少量克隆,可能错过大多数低频脱靶事件。对于体内分析,WGS 仍是首选方法,但直接捕获双链断裂 (DSB) 的技术,如 BLESS,可能比 WGS 提供更优越的能力。这是因为 WGS 还会捕获细胞/动物模型之间自然发生的变异,这可能使分析复杂化并导致误导性结果。
在 WGS 分析中,选择适当的对照组尤为重要。为了在 WGS 中最小化假阳性和假阴性,通常需要设置多个组,这既费力又昂贵。
ChIP-Seq
Cas 核酸酶,一类蛋白质,可以通过抗体拉下后进行测序,称为 ChIP-Seq。ChIP-Seq 常用于检测与 dCas9 相关的脱靶效应。与常规 Cas9 相比,常规 Cas9 会切断目标序列,导致其与 DNA 序列解离并结合不稳定,而 dCas9 因核酸酶活性受损,仍能通过 gRNA 引导结合目标序列。因此,dCas9 常用于 CRISPR 激活 (CRISPRa) 以增强基因表达,而非用于基因敲除。
DISCOVER-seq(也称为 MRE11 ChIP-Seq)是 CRISPR-Cas9 脱靶测序的最新开发方法。该方法利用内源性 DNA 修复因子 MRE11 的招募来识别整个基因组上 Cas 诱导的双链断裂。在 DNA 修复过程中,MRE11 精确地招募到 DSB 发生部位。因此,通过拉下 MRE11 结合位点并进行高通量测序,该方法允许同时检查靶向和脱靶发生。重要的是,DISCOVER-seq 适用于体内和体外样本。
Cas 核酸酶切割位点和 DSB 位点富集测序
依赖富集 Cas 核酸酶结合或切割区域的测序方法通常被称为无偏检测方法。根据这一原理,已设计了富集方法,包括:
(1) 抗体拉下:
如 ChIP-Seq,使用抗体捕获目标区域。
(2) Cas 核酸酶切割位点富集:
Digenome-Seq,简称体外 Cas9 消化全基因组测序。Cas9 切割后留下 5′ 磷酸基团,便于接头连接以进行 PCR 扩增和后续 NGS 测序。在 Digenome-Seq 中,纯化的基因组 DNA (gDNA) 在体外用 Cas9 处理,随后进行接头连接和全面全基因组测序。此方法适用于体外样本。
(3) DNA 双链断裂区域 (DSB) 富集 – DSB 捕获:
在 BLESS-Seq 中,使用直接生物素亲和富集捕获 CRISPR-Cas9 引导的 DSB 片段,随后在接头连接后进行测序。生物素亲和富集专门捕获正在发生的 DSBs,已修复的 DSBs 未被考虑,因此被标记为“脱靶快照”。此方法常用于细胞和组织样本。
在 IDLV-Seq 中,整合缺陷慢病毒载体 (IDLV) 可作为 PCR 锚点来扩增通过非同源末端连接 (NHEJ) 生成的 DSB 位点,从而检测脱靶位点。然而,此方法的效率依赖于 IDLV 通过 NHEJ 整合到 DSB 的能力。此外,由于慢病毒的随机整合特性,可能出现假阳性。
HTGST 适用于检测 CRISPR-Cas9 诱导的染色体易位,但需注意的是,CRISPR-Cas9 诱导的脱靶事件主要导致插入缺失 (indels),相对较少见。此外,此方法仍受染色质可及性限制。
与富集核酸酶切割位点的方法相比,DSB 捕获方法更具生理相关性。然而,由于大多数 DSB 持续时间极短,它们的效率可能较低。IDLV 的体内标记和原位接头连接可能受切割位点附近局部染色质和序列组成的影响。因此,捕获方法可能存在偏差。
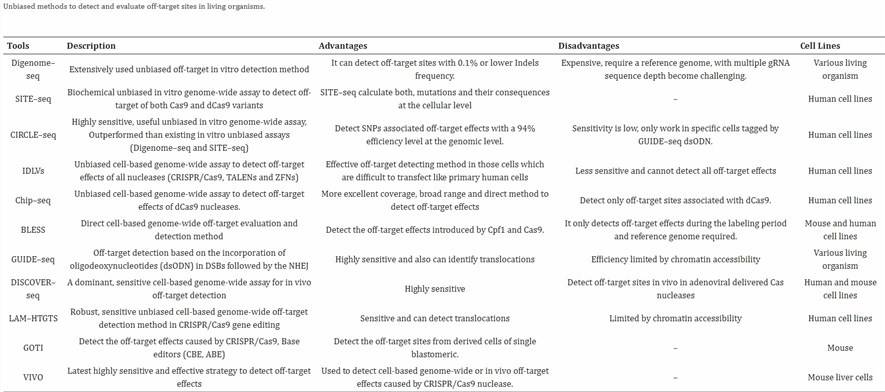
测序方法总结
多种测序方法可用,难以对每种方法进行详尽描述。以下表格提供了当前可访问的最全面总结。基于先前提供的信息,难以确定最佳方法。选择最合适的测序技术应根据实验的具体需求和研究团队的财务资源指导。
| 方法 | 适用场景 | 优点 | 局限性 |
|---|---|---|---|
| WGS | 体内分析 | 全面覆盖 | 成本高,易受自然变异干扰 |
| ChIP-Seq | dCas9 脱靶检测 | 针对性强 | 仅适用于特定蛋白结合 |
| DISCOVER-seq | 体内/体外 | 同时检测靶/脱靶 | 需要高分辨率测序 |
| Digenome-Seq | 体外样本 | 无偏检测 | 仅限体外应用 |
| BLESS-Seq | 细胞/组织 | 捕获活跃 DSB | 效率低,短时效性 |
| IDLV-Seq | NHEJ 检测 | 特定性强 | 可能有假阳性 |
| HTGST | 染色体易位 | 检测易位 | 受染色质限制 |
您可能感兴趣:
-
CRISPR 测序
-
CRISPR 筛选测序
-
CRISPR 脱靶验证
了解更多:
-
CRISPR 基因编辑评估和质量控制中的下一代测序
-
下一代测序验证您的 CRISPR/Cas9 编辑
-
CRISPR 测序:简介、工作流程和应用
CRISPR-Cas9 脱靶检测的计算模拟方法
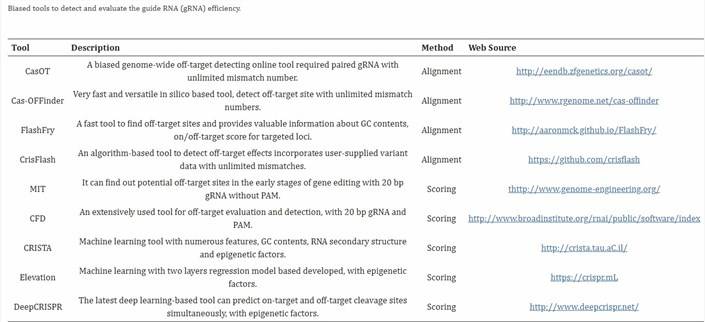
这些技术旨在预测潜在脱靶序列,主要是由于目标序列与其他序列的相似性或引导 RNA (gRNA) 的有限特异性。潜在脱靶序列可以通过整合实验数据或使用计算机算法推导。为验证脱靶效应,通常依赖 PCR 产物测序。这些方法常称为“有偏”方法,脱靶预测工具的功能可分为两种主要机制:(1) 基于序列比对的模型和 (2) 基于算法打分的预测 [1]。
CRISPR-Cas9 脱靶检测的计算模拟方法
随着 CRISPR 技术的广泛使用,其应用预期也在提高。使用 CRISPR 技术时,实验设计可能缺乏额外对照组,这可能在后续实验中带来挑战。因此,建议在 CRISPR 用于基因编辑后及时进行脱靶分析,或保留早期样本。细胞和动物均易发生自发突变。
参考文献
- Naeem, Muhammad et al. Latest Developed Strategies to Minimize the Off-Target Effects in CRISPR-Cas-Mediated Genome Editing. Cells vol. 2020
- Ipek Tasan and Huimin Zhao. Targeting Specificity of the CRISPR/Cas9 System. ACS Synthetic Biology 2017
- Wienert, B., Wyman, S.K., Yeh, C.D. et al. CRISPR off-target detection with DISCOVER-seq. Nat Protoc 15, 1775–1799 (2020)