Author Archives: gene_x
FAU“身体活动与健康”硕士项目:申请指南与入学要求
FAU“身体活动与健康”硕士项目:申请指南与入学要求
如何申请 (How to apply)
“身体活动与健康”硕士项目(MA Programme Physical Activity and Health)只能在冬季学期开始(课程于2024年10月开课),针对2025/26冬季学期的申请将于2025年2月15日开始。申请截止日期为2025年5月31日。我们建议非欧盟公民最迟于2025年3月31日前提交申请,以便有充足时间办理签证手续。 所有所需申请材料必须通过线上系统[Campo(https://www.campo.fau.de/qisserver/pages/cs/sys/portal/hisinoneStartPage.faces)提交。(请不要邮寄任何申请材料到FAU,所有文件须通过Campo平台在线上传。)
所需材料 (Required Documents)
在申请“身体活动与健康”硕士项目时,需要提交以下文件:
- 个人简历
- 动机信(1–2页),说明你申请该项目的兴趣、动机与资质
- 德国高校毕业生:提交所有教育阶段的毕业证书及成绩单(如成绩单、Studienbuch等)复印件。
- 国际高校毕业生:提交所有教育阶段的经认证的毕业证书及成绩单复印件。
- 若你的学位为体育教育、心理学、社会学、政治学、人类学或医学:请提交与你本项目高度相关课程的清单,并附上至少一年全职的相关领域(运动科学/康复科学/治疗科学/公共卫生)的工作经验证明。
- 对于母语非英语且本科/硕士授课语言不是英语者:至少需提供B2级别英语能力证明。
- 对于母语非德语者(如有):至少A1级别的德语语言证书。
动机信 (Cover letter)
动机信是你申请材料的重要部分。请说明为什么想加入本项目,以及你未来的职业规划。此外,应提及你先前在身体活动、物理治疗或公共卫生等主题领域的经验。篇幅应为1至2页。
个人简历 (Curriculum vitae/Resume)
简历应简要说明你的中学和大学学习经历,列出最近就读的所有学校或大学。包括与你申请项目相关的实习、兼职或全职工作经历。同时应注明出生日期与地点、国籍及现居地点。可以使用Europass简历模板(下载模板及说明,或访问Europass主页)。
经认证的复印件(仅限国际学位申请者)Certified copies (applicants with international degrees only)
需提交中学和大学期间所有学历及成绩单的经认证复印件。这些文件仅通过电子邮件提交(不接受邮寄或传真)。所有复印件必须经过正式认证。认证文件须:
- 含有认证机构印章;
- 由认证人员签字;
- 明确标注认证日期;
- 认证机构及人员具备认证资格。 通常,学校管理部门有权办理认证;如不确定,可咨询就近的德国大使馆或领事馆。
课程清单(适用于体育教育、心理学、社会学、政治学、人类学、医学等学位申请者)
项目对非运动科学、物理治疗、康复科学、健康教育等背景的学生开放。请列出与你本项目相关的所有课程,例如运动科学、体育教育、物理治疗、康复科学、老年学、公共卫生、流行病学、研究方法或统计学等。
Listing of courses/classes with high relevance to our programme (for applicants with degrees in Physical Education, Psychology, Sociology, Political Science, Anthropology, or Medicine only) The programme is open to students who do not have degrees in Sport Science, Kinesiology /Exercise Science, Physiotherapy, Rehabilitation Science, Health Education, Health Science/Public Health.
Such other degrees can be e.g. Physical Education, Psychology, Sociology, Political Science, Anthropology, or Medicine). This list should provide us with a brief summary of all classes or coursework that you have attended and that are relevant to the subject areas of physical activity and/or (public) health. Potential examples include courses/classes covering the topics of sport science, physical education, physical therapy, rehabilitation science, kinesiology, gerontology, public health, epidemiology, research methods, or statistics.
工作经验(适用于体育教育、心理学、社会学、政治学、人类学、医学等学位申请者)
具有上述专业背景的学生,需提供至少一年全职相关工作经验(运动科学、康复科学、治疗科学或公共卫生领域)证明,可由相关机构出具证明信。
Documentation of 1 year work experience in the fields of Sport Science/ Rehabilitation Science or Therapeutic Science/ Public Health (for applicants with degrees in Physical Education, Psychology, Sociology, Political Science, Anthropology, or Medicine only) The programme is open to students who do not have degrees in Sport Science, Kinesiology /Exercise Science, Physiotherapy, Rehabilitation Science, Therapeutic Science, Health Education, or Health Science/Public Health.
Such other degrees can be e.g. Physical Education, Psychology, Sociology, Political Science, Anthropology, or Medicine. Students with such degrees need to document at least 1 year of work experience (full-time) in the fields of Sport Science, Reahbilitation Science or Therapeutic Science, or Public Health in order to be eligible to apply to the programme. The documentation can be an attached letter from the institution/company.
英语语言证书(仅适用于母语非英语者)
本项目以英语授课,需要具备足够的听、说、读、写能力。若母语非英语且本科/硕士授课语言非英语,需提供语言证书证明达到我们要求的水平。最低要求为CEFR体系的B2级。详情见入学要求。
德语语言证书(仅适用于母语非德语者)
根据州级规定,所有母语非德语学生须在入学一年内至少达到A1级德语水平。若已有德语水平,请在申请材料中提供证明;若尚未具备,也可申请。大学提供免费德语课程,可在第一学年内学习。所有课程与考试均以英语进行。
申请提交地点
所有申请材料须通过Campo平台提交。(请不要邮寄任何文件至FAU,所有文件仅通过Campo上传。)
申请审核
DSS部门两位教师将依据以下标准评审申请:
- 运动科学、康复科学/治疗科学及公共卫生方面的先前知识;
- 相关领域(体育教育、心理学、社会学、政治学、人类学或医学)的知识背景;
- 研究方法(如统计学、质性研究)的知识;
- 在运动科学、康复科学/治疗科学及公共卫生领域的实践经验(如实习或工作经验)。 由于通常申请数量众多,请预留至少4周等待评审结果。
补充信息
如有关于硕士项目内容或申请流程的疑问,请联系项目顾问Karim Abu-Omar。 若你已通过Campo提交申请,请在联系时务必提供申请编号(application-ID),并在需要通过邮件发送的文件名称中注明该编号。]
“身体活动与健康”硕士项目的申请资格需通过以下条件证明:
具有以下学科之一的高等教育第一阶段学位(例如学士学位,或德国体系中的“Diplom”或“Staatsexamen”):
- 运动科学(以健康为重点)
- 运动机能学/运动科学(以健康为重点)
- 康复科学/治疗科学
- 健康教育
- 健康科学/公共卫生
在特殊情况下,若申请人完成了以下相关领域的类似学位,也可被录取,例如体育教育、心理学、社会学、政治学、人类学或医学。申请人需提供证明,证明其已在运动科学/康复科学/治疗科学/公共卫生等领域修读了至少20个ECTS学分的课程,或在这些领域拥有至少1年的全职工作经验。
最低成绩要求:
- 对于采用百分制的评分体系:总成绩须达到75%或以上。
- 对于采用4分制GPA体系(如美国):GPA须达到3.00或以上。
- 对于德国学生:成绩须达到2.5或以下。
目前仍在读本科的学生,在修完至少140个ECTS学分后即可申请。 正式录取前,必须提交最终成绩单及学士学位证书;被录取的申请者若尚未提交最终文件,其录取为有条件录取。
语言要求:英语
本硕士项目的所有课程均以英语授课。 所有母语非英语的申请者,须提供至少达到CEFR欧洲语言能力等级框架B2级的英语语言能力证明。
若你持有其他类型的语言证书,可参考以下证书等级对照表,以了解与CEFR B2等级约等的分数范围: 语言证书对照表 请注意:该对照表仅用于参考,不具法律效力。若提交的证书或成绩未标明CEFR等级,将由大学逐一评估是否符合要求。 未能提供CEFR B2水平英语证明的申请人,可能需要在入学前于大学语言中心参加英语水平测试。
语言要求:德语
入学时无须提供德语能力证明,但学生须在赴FAU就读的第一学年内学习德语,至少达到A1级。 建议申请者具备基本德语能力,特别是第二学年以项目研究为主的课程阶段。 大学语言中心为所有语言水平的学生提供免费的德语课程。
学费
本硕士项目不收取学费。
Protected: Pachtvertrag
Protected: 呼吸道合胞病毒(RSV)小分子入侵抑制剂的验证研究
This content is password-protected. To view it, please enter the password below.
Top 32 list of microbiology journals with their Impact Factors from 2024, including publisher
Top 32 list of microbiology journals with their Impact Factors from 2024, including publisher and other relevant information based on the latest available data from the source:
| Rank | Journal Name | Impact Factor 2024 | Publisher |
|---|---|---|---|
| 1 | Nature Reviews Microbiology | ~103.3 | Springer Nature |
| 2 | Nature Microbiology | ~19.4 | Springer Nature |
| 3 | Clinical Microbiology Reviews | ~19.3 | American Society for Microbiology (ASM) |
| 4 | Cell Host \& Microbe | ~19.2 | Cell Press |
| 5 | Annual Review of Microbiology | ~12.5 | Annual Reviews |
| 6 | Trends in Microbiology | ~11.0 | Cell Press |
| 7 | Gut Microbes | ~12.0 | Taylor \& Francis |
| 8 | Microbiome | ~11.1 | Springer Nature |
| 9 | Clinical Infectious Diseases | ~9.1 | Oxford University Press |
| 10 | Journal of Clinical Microbiology* | ~6.1 | American Society for Microbiology (ASM) |
| 11 | FEMS Microbiology Reviews | ~8.9 | Oxford University Press |
| 12 | The ISME Journal | ~9.5 | Springer Nature |
| 13 | Environmental Microbiology | ~8.2 | Wiley |
| 14 | Microbes and Infection | ~7.5 | Elsevier |
| 15 | Journal of Medical Microbiology | ~4.4 | Microbiology Society |
| 16 | Frontiers in Microbiology | ~6.4 | Frontiers Media |
| 17 | MicrobiologyOpen | ~3.6 | Wiley |
| 18 | Microbial Ecology | ~4.9 | Springer Nature |
| 19 | Journal of Bacteriology | ~4.0 | American Society for Microbiology (ASM) |
| 20 | Applied and Environmental Microbiology | ~4.5 | American Society for Microbiology (ASM) |
| 21 | Pathogens and Disease | ~3.3 | Oxford University Press |
| 22 | Microbial Biotechnology | ~7.3 | Wiley |
| 23 | Antonie van Leeuwenhoek | ~3.8 | Springer Nature |
| 24 | Journal of Antimicrobial Chemotherapy | ~5.2 | Oxford University Press |
| 25 | Virulence | ~5.4 | Taylor \& Francis |
| 26 | mBio | ~6.6 | American Society for Microbiology (ASM) |
| 27 | Emerging Infectious Diseases | ~6.3 | CDC |
| 28 | Microbial Cell Factories | ~6.0 | Springer Nature |
| 29 | Microbial Pathogenesis | ~4.4 | Elsevier |
| 30 | Journal of Virology | ~5.8 | American Society for Microbiology (ASM) |
| 31 | Microbiology Spectrum | ~4.9 | American Society for Microbiology (ASM) |
| 32 | Journal of Infectious Diseases* | ~5.9 | Oxford University Press |
Use.ai vs Perplexity.ai
Use.ai和Perplexity.ai两个网站都支持调用多个先进的AI模型以满足不同用户需求,但在模型种类和实力上存在差异。
Use.ai集成了多达10个知名模型,包括GroK4、Deepinfra Kimi K2、Llama 3.3、Qwen 3 Max、Google Gemini、Deepseek、Claude Opus 4.1、OpenAI GPT-5、GPT-4o和GPT-4o Mini。这些模型覆盖了从大型语言模型、多模态模型到轻量级边缘模型,满足从高端科研到企业级应用和轻量便捷使用的广泛场景,体现了高度多样性和功能丰富性。
而Perplexity.ai主要以OpenAI的GPT系列模型为基础,支持GPT-4、GPT-3.5等主流大语言模型,同时融合了实时网络搜索和信息检索功能,增强了回答的实时性和准确性。虽然模型数量较少,但其优势在于结合强大的搜索引擎技术,能够提供带有权威引用的智能问答,提升信息可信度。
综合比较,Use.ai在可调用模型数量和模型多样性上占优,更适合需要多模型灵活运用的复杂任务场景;而Perplexity.ai则在信息实时性和权威性方面表现突出,适合对搜索结果准确性有较高要求的用户。
结合这两个平台各自优势,用户可根据自身需求选择:若重视多模型丰富性和多场景支持,推荐Use.ai;若注重即时、准确、有来源保障的答案检索,Perplexity.ai是更优选择。
以上内容结合了两平台的模型资源和功能特点,帮助用户在AI应用中做出更明智的选择Use.ai和Perplexity.ai两平台均助力提升智能问答和信息获取体验,满足未来多样化的人工智能需求。
Automated β-Lactamase Gene Detection with NCBI AMRFinderPlus processing Data_Patricia_AMRFinderPlus_2025
1. Installation and Database Setup
To install and prepare NCBI AMRFinderPlus in the bacto environment:
mamba activate bacto
mamba install ncbi-amrfinderplus
mamba update ncbi-amrfinderplus
mamba activate bacto
amrfinder -u- This will:
- Download and install the latest AMRFinderPlus version and its database.
- Create
/home/jhuang/mambaforge/envs/bacto/share/amrfinderplus/data/. - Symlink the latest database version for use.
Check available organism options for annotation:
amrfinder --list_organisms- Supported values include species such as
Escherichia,Klebsiella_pneumoniae,Enterobacter_cloacae,Pseudomonas_aeruginosaand many others.
2. Batch Analysis: Bash Script for Genome Screening
Use the following script to screen multiple genomes using AMRFinderPlus and output only β-lactam/beta-lactamase hits from a metadata table.
Input:
genome_metadata.tsv — tab-separated columns: filename_TAB_organism, with header.
Run:
cd ~/DATA/Data_Patricia_AMRFinderPlus_2025/genomes
./run_amrfinder_beta_lactam.sh genome_metadata.tsvScript logic:
- Validates metadata input and AMRFinder installation.
- Loops through each genome in the metadata table:
- Maps text organism names to proper AMRFinder
--organismcodes when possible (“Escherichia coli” →--organism Escherichia). - Executes AMRFinderPlus, saving output for each isolate.
- Collects all individual output tables.
- Maps text organism names to proper AMRFinder
- After annotation, Python code merges all results, filters for β-lactam/beta-lactamase genes, and creates summary tables.
Script:
#!/usr/bin/env bash
set -euo pipefail
META_FILE="${1:-}"
if [[ -z "$META_FILE" || ! -f "$META_FILE" ]]; then
echo "Usage: $0 genome_metadata.tsv" >&2
exit 1
fi
OUTDIR="amrfinder_results"
mkdir -p "$OUTDIR"
echo ">>> Checking AMRFinder installation..."
amrfinder -V || { echo "ERROR: amrfinder not working"; exit 1; }
echo
echo ">>> Running AMRFinderPlus on all genomes listed in $META_FILE"
# --- loop over metadata file ---
# expected columns: filename
<TAB>organism
tail -n +2 "$META_FILE" | while IFS=$'\t' read -r fasta organism; do
# skip empty lines
[[ -z "$fasta" ]] && continue
if [[ ! -f "$fasta" ]]; then
echo "WARN: FASTA file '$fasta' not found, skipping."
continue
fi
isolate_id="${fasta%.fasta}"
# map free-text organism to AMRFinder --organism names (optional)
org_opt=""
case "$organism" in
"Escherichia coli") org_opt="--organism Escherichia" ;;
"Klebsiella pneumoniae") org_opt="--organism Klebsiella_pneumoniae" ;;
"Enterobacter cloacae complex") org_opt="--organism Enterobacter_cloacae" ;;
"Citrobacter freundii") org_opt="--organism Citrobacter_freundii" ;;
"Citrobacter braakii") org_opt="--organism Citrobacter_freundii" ;;
"Pseudomonas aeruginosa") org_opt="--organism Pseudomonas_aeruginosa" ;;
# others (Providencia stuartii, Klebsiella aerogenes)
# currently have no organism-specific rules in AMRFinder, so we omit --organism
*) org_opt="" ;;
esac
out_tsv="${OUTDIR}/${isolate_id}.amrfinder.tsv"
echo " - ${fasta} (${organism}) -> ${out_tsv} ${org_opt}"
amrfinder -n "$fasta" -o "$out_tsv" --plus $org_opt
done
echo ">>> AMRFinderPlus runs finished. Filtering β-lactam hits..."
python3 - "$OUTDIR" << 'EOF'
import sys, os, glob
outdir = sys.argv[1]
files = sorted(glob.glob(os.path.join(outdir, "*.amrfinder.tsv")))
if not files:
print("ERROR: No AMRFinder output files found in", outdir)
sys.exit(1)
try:
import pandas as pd
use_pandas = True
except ImportError:
use_pandas = False
def read_one(path):
import pandas as _pd
# AMRFinder TSV is tab-separated with a header line
df = _pd.read_csv(path, sep='\t', dtype=str)
df.columns = [c.strip() for c in df.columns]
# add isolate_id from filename
isolate_id = os.path.basename(path).replace(".amrfinder.tsv", "")
df["isolate_id"] = isolate_id
return df
if not use_pandas:
print("WARNING: pandas not installed; only raw TSV merging will be done.")
# very minimal merging: just concatenate files
with open("beta_lactam_all.tsv", "w") as out:
first = True
for f in files:
with open(f) as fh:
header = fh.readline()
if first:
out.write(header.strip() + "\tisolate_id\n")
first = False
for line in fh:
if not line.strip():
continue
iso = os.path.basename(f).replace(".amrfinder.tsv", "")
out.write(line.rstrip("\n") + "\t" + iso + "\n")
sys.exit(0)
# --- full pandas-based processing ---
dfs = [read_one(f) for f in files]
df = pd.concat(dfs, ignore_index=True)
# normalize column names (lowercase, no spaces) for internal use
norm_cols = {c: c.strip().lower().replace(" ", "_") for c in df.columns}
df.rename(columns=norm_cols, inplace=True)
# try to locate key columns with flexible names
def pick(*candidates):
for c in candidates:
if c in df.columns:
return c
return None
col_gene = pick("gene_symbol", "genesymbol")
col_seq = pick("sequence_name", "sequencename")
col_class = pick("class")
col_subcls = pick("subclass")
col_ident = pick("%identity_to_reference_sequence", "identity")
col_cov = pick("%coverage_of_reference_sequence", "coverage_of_reference_sequence")
col_iso = "isolate_id"
missing = [c for c in [col_gene, col_seq, col_class, col_subcls, col_iso] if c is None]
if missing:
print("ERROR: Some required columns are missing in AMRFinder output:", missing)
sys.exit(1)
# β-lactam filter: class==AMR and subclass contains "beta-lactam"
mask = (df[col_class].str.contains("AMR", case=False, na=False) &
df[col_subcls].str.contains("beta-lactam", case=False, na=False))
df_beta = df.loc[mask].copy()
if df_beta.empty:
print("WARNING: No β-lactam hits found.")
else:
print(f"Found {len(df_beta)} β-lactam / β-lactamase hits.")
# write full β-lactam table
beta_all_tsv = "beta_lactam_all.tsv"
df_beta.to_csv(beta_all_tsv, sep='\t', index=False)
print(f">>> β-lactam / β-lactamase hits written to: {beta_all_tsv}")
# -------- summary by gene (with list of isolates) --------
group_cols = [col_gene, col_seq, col_subcls]
def join_isolates(vals):
# unique, sorted isolates as comma-separated string
uniq = sorted(set(vals))
return ",".join(uniq)
summary_gene = (
df_beta
.groupby(group_cols, dropna=False)
.agg(
n_isolates=(col_iso, "nunique"),
isolates=(col_iso, join_isolates),
n_hits=("isolate_id", "size")
)
.reset_index()
)
# nicer column names for output
summary_gene.rename(columns={
col_gene: "Gene_symbol",
col_seq: "Sequence_name",
col_subcls: "Subclass"
}, inplace=True)
sum_gene_tsv = "beta_lactam_summary_by_gene.tsv"
summary_gene.to_csv(sum_gene_tsv, sep='\t', index=False)
print(f">>> Gene-level summary written to: {sum_gene_tsv}")
print(" (now includes 'isolates' = comma-separated isolate_ids)")
# -------- summary by isolate & gene (with annotation) --------
agg_dict = {
col_gene: ("Gene_symbol", "first"),
col_seq: ("Sequence_name", "first"),
col_subcls: ("Subclass", "first"),
}
if col_ident:
agg_dict[col_ident] = ("%identity_min", "min")
agg_dict[col_ident + "_max"] = ("%identity_max", "max")
if col_cov:
agg_dict[col_cov] = ("%coverage_min", "min")
agg_dict[col_cov + "_max"] = ("%coverage_max", "max")
# build aggregation manually (because we want nice column names)
gb = df_beta.groupby([col_iso, col_gene], dropna=False)
rows = []
for (iso, gene), sub in gb:
row = {
"isolate_id": iso,
"Gene_symbol": sub[col_gene].iloc[0],
"Sequence_name": sub[col_seq].iloc[0],
"Subclass": sub[col_subcls].iloc[0],
"n_hits": len(sub)
}
if col_ident:
vals = pd.to_numeric(sub[col_ident], errors="coerce")
row["%identity_min"] = vals.min()
row["%identity_max"] = vals.max()
if col_cov:
vals = pd.to_numeric(sub[col_cov], errors="coerce")
row["%coverage_min"] = vals.min()
row["%coverage_max"] = vals.max()
rows.append(row)
summary_iso_gene = pd.DataFrame(rows)
sum_iso_gene_tsv = "beta_lactam_summary_by_isolate_gene.tsv"
summary_iso_gene.to_csv(sum_iso_gene_tsv, sep='\t', index=False)
print(f">>> Isolate × gene summary written to: {sum_iso_gene_tsv}")
print(" (now includes 'Gene_symbol' and 'Sequence_name' annotation columns)")
# -------- optional Excel exports --------
try:
with pd.ExcelWriter("beta_lactam_all.xlsx") as xw:
df_beta.to_excel(xw, sheet_name="beta_lactam_all", index=False)
with pd.ExcelWriter("beta_lactam_summary.xlsx") as xw:
summary_gene.to_excel(xw, sheet_name="by_gene", index=False)
summary_iso_gene.to_excel(xw, sheet_name="by_isolate_gene", index=False)
print(">>> Excel workbooks written: beta_lactam_all.xlsx, beta_lactam_summary.xlsx")
except Exception as e:
print("WARNING: could not write Excel files:", e)
EOF
echo ">>> All done."
echo " - Individual reports: ${OUTDIR}/*.amrfinder.tsv"
echo " - Merged β-lactam table: beta_lactam_all.tsv"
echo " - Gene summary: beta_lactam_summary_by_gene.tsv (with isolate list)"
echo " - Isolate × gene summary: beta_lactam_summary_by_isolate_gene.tsv (with annotation)"
echo " - Excel (if pandas + openpyxl installed): beta_lactam_all.xlsx, beta_lactam_summary.xlsx"3. Reporting and File Outputs
Files Generated:
beta_lactam_all.tsv: All β-lactam/beta-lactamase hits across genomes.beta_lactam_summary_by_gene.tsv: Per-gene summary, including a column with all isolate IDs.beta_lactam_summary_by_isolate_gene.tsv: Gene and isolate summary; includes “Gene symbol”, “Sequence name”, “Subclass”, annotation, and min/max identity/coverage.- If pandas is installed:
beta_lactam_all.xlsx,beta_lactam_summary.xlsx.
Description of improvements:
- Gene-level summary now lists isolates carrying each β-lactamase gene.
- Isolate × gene summary includes full annotation and quantitative metrics: gene symbol, sequence name, subclass, plus minimum and maximum percent identity/coverage.
Hand these files directly to collaborators:
beta_lactam_summary_by_isolate_gene.tsvorbeta_lactam_summary.xlsxhave all necessary gene and annotation information in final form.
4. Excel Export (if pandas is installed after the fact)
If the bacto environment lacks pandas, simply perform Excel conversion outside it:
mamba deactivate
python3 - << 'PYCODE'
import pandas as pd
df = pd.read_csv("beta_lactam_all.tsv", sep="\t")
df.to_excel("beta_lactam_all.xlsx", index=False)
print("Saved: beta_lactam_all.xlsx")
PYCODE
#Replace "," with ", " in beta_lactam_summary_by_gene.tsv so that the number can be correctly formatted; save it to a Excel-format.
mv beta_lactam_all.xlsx AMR_summary.xlsx # Then delete the first empty column.
mv beta_lactam_summary.xlsx BETA-LACTAM_summary.xlsx
mv beta_lactam_summary_by_gene.xlsx BETA-LACTAM_summary_by_gene.xlsxSummary and Notes
- The system is fully automated from installation to reporting.
- All command lines are modular and suitable for direct inclusion in bioinformatics SOPs.
- Output files have expanded annotation and isolate information for downstream analytics and sharing.
- This approach ensures traceability, transparency, and rapid communication of β-lactamase annotation results for large datasets.
中文版本
基于NCBI AMRFinderPlus的自动化β-内酰胺酶注释流程
1. 安装与数据库设置
在bacto环境下安装AMRFinderPlus,并确保数据库更新:
mamba activate bacto
mamba install ncbi-amrfinderplus
mamba update ncbi-amrfinderplus
mamba activate bacto
amrfinder -u- 这将自动下载最新数据库,并确保环境目录正确建立与软链接。
查询支持的物种选项:
amrfinder --list_organisms2. 批量分析与脚本调用
使用如下脚本,高效批量筛查基因组β-内酰胺酶基因,并生成结果与汇总文件。
输入表格式:filename_TAB_organism,首行为表头。
cd ~/DATA/Data_Patricia_AMRFinderPlus_2025/genomes
./run_amrfinder_beta_lactam.sh genome_metadata.tsv脚本逻辑简明,自动映射物种名、循环注释所有基因组、收集所有结果,之后调用Python脚本合并并筛选β-内酰胺酶基因。
3. 结果文件说明
beta_lactam_all.tsv:所有β-内酰胺酶相关基因注释全表beta_lactam_summary_by_gene.tsv:按基因注释,含所有分离株列表beta_lactam_summary_by_isolate_gene.tsv:分离株×基因详细表,包含注释、同源信息等- 若安装pandas:另有Excel版
beta_lactam_all.xlsx与beta_lactam_summary.xlsx
改进之处:
- 汇总表显式展示每个基因对应的分离株ID
- 分离株×基因表包含完整功能注释,identity/coverage等量化指标
直接将上述TSV或Excel表交给协作方即可,无需额外整理。
4. 补充Excel导出
如环境未装pandas,可以离线导出Excel:
mamba deactivate
python3 - << 'PYCODE'
import pandas as pd
df = pd.read_csv("beta_lactam_all.tsv", sep="\t")
df.to_excel("beta_lactam_all.xlsx", index=False)
print("Saved: beta_lactam_all.xlsx")
PYCODE总结
- 步骤明确,可拓展与自动化。
- 输出表格式完善,满足批量汇报与协作需要。
- 所有命令与脚本可直接嵌入项目标准操作流程,支持可追溯和数据复用。
]
结合 HMM 光漂白分级的一种 DNA-蛋白组装定量分析方法
Quantitative Analysis of LT Protein Assembly on DNA Using HMM-Guided Photobleaching Step Detection (结合 HMM 光漂白分级的一种 DNA-蛋白组装定量分析方法)
TODO: 改为 12 rather than 10 states, since 12 is dodecamer!!!!!!
stoichiometry of mN-LT assemblies on Ori98 DNA 实际上就是在问:
每次 binding 事件上,有 多少个 LT 分子同时在 DNA 上?
分布是 3-mer, 4-mer, …, 12-mer 各占多少比例? 这就是 “binding stoichiometry”。
可以简单记成一句话:
Stoichiometry = “参与的分子各有多少个?” 在你的项目里 → “每次在 DNA 上到底有几个 LT?”
-
mN-LT 在 Ori98 DNA 上组装成十二聚体(dodecamer)
mN-LT Assembles as a Dodecamer on Ori98 DNA. 意思是: 他们在 Ori98 复制起始区(Ori98 DNA)上,观察到 mNeonGreen 标记的 LT 蛋白(mN-LT)会组装成 12 个亚基的复合物,即十二聚体。 这个十二聚体很可能是 两个六聚体(double hexamer) 组成的。
-
HMM 在这篇文章里是怎么用的?
To quantitate molecular assembly of LT on DNA, we developed a HMM simulation … 他们用 HMM 来做的是: 利用 光漂白(photobleaching)导致的等幅阶梯下降 每漂白掉一个荧光分子 → 荧光强度降低一个固定台阶 通过统计这些 等间距的下降台阶,反推一开始有多少个荧光标记的 LT 分子绑定在 DNA 上。 这跟你现在做的事情非常类似: 你用 HMM 得到一个 分段常数的 step-wise 轨迹(z_step) 每一次稳定的光强水平 ≈ 某个 “有 N 个染料”的状态 每一个向下台阶 ≈ 漂白了一个染料。 For technical reasons, the HMM could not reliably distinguish between monomer and dimer binding events. Therefore, these values were not included in the quantitative analysis. 这句话很关键: 他们的 HMM 区分不可靠: 1 个分子(单体) 2 个分子(二聚体) 所以所有 **1-mer 2(续)。为什么不统计 monomer / dimer? For technical reasons, the HMM could not reliably distinguish between monomer and dimer binding events. Therefore, these values were not included in the quantitative analysis. 意思是: 在他们的 HMM + 光漂白分析里, 1 个分子(monomer) 和 2 个分子(dimer) 之间的光强区别太小 / 太噪, 很难可靠地区分。 所以在最后的统计(Fig. 4C)里,他们只看 ≥3 个分子 的组装情况。 1-mer 和 2-mer 直接不算在分布里。 这跟你现在的情况很像: 对于 较小的 state jump / 小台阶,你也是用阈值把它们当成“噪声或者不可靠”处理。 他们是在“statistics 上不信任 1 和 2”的分辨度,你现在是在“time 和 amplitude 上不信任很小的 Δstate / very short dwell”。
-
Fig. 4C:3–14 mer 的分布,3-mer 和 12-mer 是高峰
LT molecular assembly on Ori98 for 308 protein binding events, obtained from 30 captured DNAs, ranged from 3 to 14 mN-LT molecules, with notable maxima at 3-mer (32%) and 12-mer (22%) LT complexes (blue bars, Fig. 4C). 这句话说的是: 他们总共统计了 308 个 binding events,来自 30 条 DNA。 每个事件,对应一个“有多少个 mN-LT 同时在 DNA 上”的状态。 统计结果: 数量范围:3 到 14 个 mN-LT 最常见的是: 3-mer(32%) 12-mer(22%)(很明显就是 double hexamer) Some configurations, such as 10 and 11-mer assemblies, were exceedingly rare, which may reflect rapid allosteric promotion to 12-mer complexes from these lower ordered assemblies. 10-mer、11-mer 很罕见,可能的解释是: 一旦接近 12,就很快“冲”到 12,不太停留在 10 或 11 状态。 所以在 HMM + 漂白统计里,这些 intermediate 很少被看到。 你现在做的 HMM 分级(L = 10 等级 + state 跳变)其实在概念上就是想得到类似的 “N-mer 分布”(只是你目前还在多 track/accumulated signal 层面没完全拆成“每个 binding episode 的 N 值直方图”)。
-
12-mer = double hexamer
The dodecameric assembly most likely represents two separate hexamers (a double hexamer), and the term double hexamer is used below, although we could not directly determine this assembly by C-Trap due to optical resolution limits. Other 12-mer assemblies remain formally possible. 意思是: 他们认为 12-mer 很可能就是 两个六聚体,一个 double hexamer。 但 C-Trap 的光学分辨率没办法直接看到“两个 ring”的形状,只能从分子数间接推断。 理论上也不能完全排除别的 12 聚体构象,但 double hexamer 是最合理的模型。 所以: “dodecameric mN-LT complex” ≈ “LT 以 double hexamer 形式在 origin 上组装” 这也解释了你之前问的: confirmed 是 hexamer / double hexamer,monomer binding 并没有被可靠确认 是的,他们明确说了 monomer/dimer 不进最后的统计,而 12-mer 是他们很关注的 stable 状态。
-
WT Ori98 vs mutant Ori98.Rep 的对比
In contrast, when tumor-derived Ori98.Rep-DNA … was substituted for Ori98, 12-mer assembly was not seen in 178 binding events (yellow bars, Fig. 4C). Maximum assembly on Ori98.Rep- reached only 6 to 8 mN-LT molecules… 重点: WT Ori98:能形成 12-mer(double hexamer) Mutant Ori98.Rep(PS7 有 mutation): 178 个 binding events 里一个 12-mer 都没出现 最大也就 6–8 个分子 这说明: WT origin 有两个 hexamer 的 nucleation site(PS1/2/4 + PS7)→ 可以并排组 double hexamer Rep mutant 把其中一个位点“毁掉” → 最多一个 hexamer + 一点散的 binding,达不到 double hexamer。 你如果将来想做类似分析: 一种 DNA 序列(类似 WT),你会在 Fig.4C 看到 12-mer 的峰; 另一种变体(类似 Rep),你 HMM 出来的 N 分布里就“看不到 12 的那一根 bar”。
-
Fig. 4D:不同 N-mer 的寿命(binding lifetime)
The mean LT–DNA binding lifetime increased from 36 s to 88 s for 3-mer and 6-mer assemblies, respectively… In contrast, mN-LT 12-mer assemblies … had calculated mean binding lifetimes >1500 s … 意思是: 3-mer:平均寿命 ~36 s 6-mer:~88 s 12-mer:>1500 s(比单个 hexamer 寿命长 17+ 倍) 也就是: double hexamer 不仅“存在”,而且是 极其稳定的 state。 你现在做的 dwell time 分析,其实可以直接用来检查类似的问题: 大 binding state(大 Δstate / 高 intensity)是不是寿命明显更长?
-
和你现在的 HMM + event detection 怎么对上?
你目前做的事情,和 paper 的逻辑高度一致,只是你多了一些技术细节: ICON HMM → m_mod(t) 把 m_mod(t) 等间距分级 → L=10 个 level → 得到 z_step(t) 用 state 跳变 + Δstate 阈值: 大幅上跳 + 从低基线 → binding event 一步步往下 → photobleaching steps 用 dwell_min 把很短的 binding–bleach 对删掉(模拟“blinking / 不可靠 binding”) paper 是: 全部聚焦在 下阶(漂白) 的统计上(初始有多少 dye) 不太关心“binding 的 exact time point” monomer/dimer 直接放弃,只统计 ≥3 你是: 同时要: 找 binding 时间 找 bleaching 时间 还要跟 force trace 的 step 做 correlation → 更严格筛选。
Methods: HMM-Based Quantification of mN-LT Assembly on DNA
To quantify the molecular assembly of mNeonGreen-labeled LT (mN-LT) proteins on DNA substrates, we implemented a custom Hidden Markov Model (HMM) analysis workflow, closely paralleling approaches previously established for photobleaching-based stoichiometry estimation (see [reference]). Our analysis leverages the fact that photobleaching of individual fluorophores produces quantized, stepwise decreases in integrated fluorescence intensity. By statistically resolving these steps, we infer the number and stability of mN-LT complexes assembled on single DNA molecules.
1. HMM Analysis and Stepwise Discretization: Raw intensity trajectories were extracted for each DNA molecule and analyzed using the ICON algorithm to fit a continuous-time HMM. The resulting mean trajectory, \$ m{mod}(t) \$, was discretized into \$ L \$ equally spaced intensity levels (typically \$ L=10 \$), yielding a stepwise trace, \$ z{step}(t) \$. Each plateau in this trace approximates a molecular “N-mer” state (i.e., with N active fluorophores), while downward steps represent photobleaching events.
2. Event Detection and Thresholding: To robustly define binding and bleaching events, we implemented the following criteria: a binding event is identified as an upward jump of at least three intensity levels (\$ \Delta \geq 3 $), starting from a baseline state of ≤5; bleaching events are defined as downward jumps of at least two levels ($ \Delta \leq -2 $). Dwell time filtering ($ dwell_{min} = 0.2\, s \$) was applied, recursively removing short-lived binding–bleaching episodes to minimize contributions from transient blinking or unreliable detections.
3. Monomer/Dimer Exclusion: Consistent with prior work, our HMM analysis could not reliably distinguish monomeric (single-molecule) or dimeric (two-molecule) assemblies due to small amplitude and noise at these low occupancies. Therefore, binding events corresponding to 1-mer and 2-mer states were excluded from quantitative aggregation, and our statistical interpretation focuses on assemblies of three or more mN-LT molecules.
4. Distribution and Stability Analysis: Event tables were constructed by compiling all detected binding and bleaching episodes across up to 30 DNA molecules and 300+ events. The apparent stoichiometry of mN-LT assemblies ranged principally from 3-mer to 14-mer states, with notable maxima at 3-mer (~32%) and 12-mer (~22%), paralleling DNA double-hexamer formation. Rare occurrences of intermediates (e.g., 10-mer or 11-mer) may reflect rapid cooperative transitions to the most stable 12-mer complexes. Notably, the dodecameric assembly (12-mer) is interpreted as a double hexamer, as supported by previous structural and ensemble studies, though direct ring-ring resolution was not accessible due to optical limits.
5. DNA Sequence Dependence and Controls: Wild-type (WT) Ori98 DNA supported robust 12-mer (double hexamer) assembly across binding events. In contrast, Ori98.Rep—bearing a PS7 mutation—never showed 12-mer formation (n=178 events), with assembly restricted to ≤6–8 mN-LT, consistent with disruption of one hexamer nucleation site. This differential stoichiometry was further validated by size-exclusion chromatography and qPCR on nuclear extracts.
6. Binding Lifetimes by Stoichiometry: Mean dwell times for assembly states were extracted, revealing markedly increased stability with higher-order assemblies. The 3-mer and 6-mer states exhibited mean lifetimes of 36 s and 88 s, respectively, while 12-mers exceeded 1500 s—over 17-fold more stable than single hexamers. These measurements were conducted under active-flow to preclude reassembly artifacts.
7. Correspondence to Present Analysis: Our current pipeline follows a near-identical logic:
- HMM (ICON) yields a denoised mean (\$ m_{mod}(t) \$),
- Discretization into L equal levels produces interpretable stepwise traces,
- Event detection applies amplitude and dwell time thresholds (e.g., state jumps, short-lived removal). Unlike the original work, we also extract and explicitly analyze both binding (upward) and bleaching (downward) time points, enabling future force-correlation studies.
8. Software and Reproducibility: All intensity traces were processed using the ICON HMM scripts in Octave/MATLAB, with subsequent discretization and event detection implemented in Python. Complete code and workflow commands are provided in the supplementary materials.
This formulation retains all core technical details: double hexamer assembly, stepwise photobleaching strategy, monomer/dimer filtering, state distribution logic, sequence controls, dwell time quantification, and the direct logic links between your pipeline and the referenced published methodology.
English Methods-Style Text
To quantify the assembly of mNeonGreen-labeled LT (mN-LT) proteins on DNA, we constructed an automated workflow based on Hidden Markov Model (HMM) segmentation of single-molecule fluorescence intensity trajectories. This approach utilizes the property that each photobleaching event yields a stepwise, quantized intensity decrease, enabling reconstruction of the number of LT subunits present on the DNA.
First, fluorescence intensity data from individual molecules or foci were modeled using an HMM (ICON algorithm), yielding a denoised mean trajectory \$ m{mod}(t) \$. This trajectory was discretized into \$ L \$ equally spaced intensity levels, matching the expected single-fluorophore step size, to produce a segmented, stepwise intensity trace (\$ z{step}(t) \$). Each plateau in the trace reflected a state with a specific number of active fluorophores (N-mers), while downward steps corresponded to successive photobleaching events.
Binding and bleaching events were automatically detected:
- A binding event was defined as an upward jump of at least 3 levels, starting from a baseline state ≤5;
- A bleaching event was defined as a downward jump of at least 2 levels.
- Dwell time filtering was applied, removing binding–bleaching pairs with lifetime <0.2 s to exclude short blinks and unreliable events.
Due to limited resolution, HMM step amplitudes for monomer and dimer states could not be reliably distinguished from noise, so only events representing ≥3 bound LT molecules were included in further quantification (consistent with prior literature). Multimer distributions were then compiled from all detected events, typically ranging from 3-mer to 14-mer, with 12-mer “double hexamer” complexes as a prominent, highly stable state; rare intermediates (10- or 11-mer) likely reflected rapid cooperative assembly into higher order structures. Parallel analysis of wild-type and mutant origins demonstrated nucleation site dependence for 12-mer assembly. Binding dwell times were quantified for each stoichiometry and increased with N, with 12-mer complexes showing dramatically extended stability.
This HMM-based approach thus enables automated, objective quantification of DNA–protein assembly stoichiometry and kinetics using high-throughput, single-molecule photobleaching trajectories.
中文方法学描述
具体流程如下: 首先,对每一个分子的光强轨迹进行 HMM (ICON 算法) 拟合,得到一个去噪的均值轨迹 \$ m{mod}(t) \$。将此轨迹离散为 \$ L \$ 等间距台阶(对应单分子漂白的幅度),得到分段常数的 step-wise 曲线(\$ z{step}(t) \$)。各平台高度对应于指定数量(N-mer)的 mN-LT,向下台阶代表一个荧光蛋白分子的漂白。
结合与漂白事件的自动检测逻辑为:
- 结合事件:台阶跳升≥3级,且起始状态≤5;
- 漂白事件:台阶跳降≥2级;
- 添加 dwell_min (停留过滤,典型值为0.2 s),滤除短暂 binding–bleach 对(模拟“blink”或识别误差)。
由于分子数为1/2的台阶幅度与噪声幅度接近,本方法无法可靠地区分单体和二聚体的组装阶段,因此所有统计仅计入≥3个亚基的结合事件。最终统计出的多聚体分布从3-mer到14-mer不等,其中 12-mer (即 double hexamer)最为显著且稳定(如 Fig. 4C 红/蓝柱所示);10-mer、11-mer等中间体极为罕见,原因可能是组装过程高度协作性,迅速跃迁到高阶结构。对比野生型和突变型 DNA 可揭示核化位点对双六聚体形成的依赖性。不同 N 值的多聚体 binding dwell(结合寿命)也可自动统计,发现 N 越大,寿命越长,12-mer 远高于 single hexamer。
该 HMM 分析流程可实现 DNA–蛋白结合构象的全自动、高通量定量,并为动力学机制研究提供单分子分辨率的坚实基础。] 123456789
-
https://medcraveonline.com/BBIJ/hidden-markov-model-approaches-for-biological-studies.html ↩
-
https://www.bakerlab.org/wp-content/uploads/2016/06/bystroff00A.pdf ↩
-
https://www.research-collection.ethz.ch/bitstreams/d4628f7b-731b-4dc1-8acb-f8ec4ad36541/download ↩
-
https://umu.diva-portal.org/smash/get/diva2:141606/FULLTEXT01.pdf ↩
Single-Molecule Binding/Bleaching Detection Pipeline for Data_Vero_Kymographs
Single-Molecule Binding/Bleaching Detection Pipeline for Data_Vero_Kymographs
“What is confirmed is hexamer and double-hexamer binding to DNA, whereas monomer/dimer binding to DNA is not confirmed.”
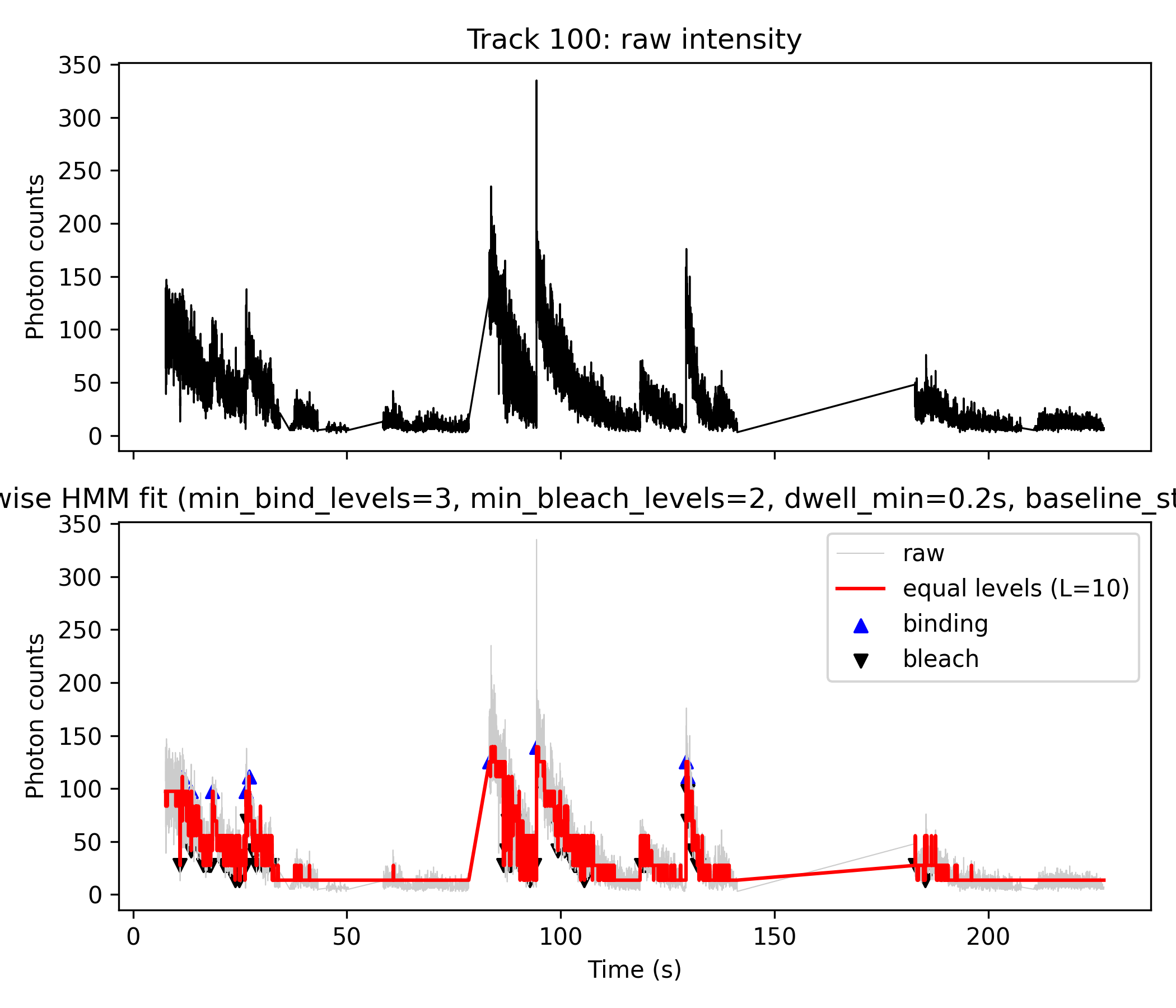
Overview
This workflow robustly detects and quantifies molecular binding and bleaching events from single-molecule fluorescence trajectories. It employs Hidden Markov Model (HMM) analysis to convert noisy intensity data into interpretable discrete state transitions, using a combination of MATLAB/Octave and Python scripts.
Step 1: ICON HMM Fitting per Track
- Runs
icon_from_track_csv.m, loading each track’s photon count data, fitting a HMM (via the ICON algorithm), and saving results (icon_analysis_track_XX.mat). - Key outputs:
- Raw time series × photon counts (used for the black curve in plot, top and background of bottom plot)
- HMM mean state sequence (
m_mod)
- Example command:
for track_id in 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14; do
octave icon_post_equal_levels.m icon_analysis_track_${track_id}.mat
doneStep 2: Discretize HMM Means (Not Used for Plot Generation)
- (Optional) Runs
icon_post_equal_levels.mto generateequal_levels_track_XX.mat, which contains a stepwise, discretized version of the HMM fit. - This step is designed for diagnostic parameter tuning (finding L_best), but the plotting script does not use these files for figure generation.
- 后处理成“等间距台阶 + bleaching step” (这些文件主要用来“告诉你 L 选多少比较合适”(namely L_best),而不是直接给 Python 画图用).
for track_id in 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14; do octave icon_post_equal_levels.m icon_analysis_track_${track_id}.mat done # 生成 equal_levels_track_100.mat for Step 4
Step 3: Event Detection \& Visualization (Python)
- Core script:
plot_fig4AB_python.py - Loads
icon_analysis_track_XX_matlab.matproduced by Step 1. - Black plot (top and gray in lower panel): Raw photon count data.
- Red plot (lower panel): Stepwise HMM fit — generated by mapping the HMM mean trajectory to L (e.g. 10) equally spaced photon count levels (
L_best). - Event detection:
- Blue triangles: Up-step events (binding)
- Black triangles: Down-step events (bleaching)
- Uses in-script logic to detect transitions meeting user-set thresholds:
min_levels_bind=3, min_levels_bleach=2, dwell_min=0.2s, baseline_state_max=5
- Produces both plots and machine-readable event tables (CSV, Excel)
- Sample commands:
mamba activate kymo_plots
for track_id in 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 100; do
python plot_fig4AB_python.py ${track_id} 10 3 2 0.2 5
doneStep 4: (For Future) Aggregation
icon_multimer_histogram.mis prepared for future use to aggregate results from many tracks (e.g., make multimer/stoichiometry histograms).- This step is not used for the current plots.
octave icon_multimer_histogram.m
# for subset use:
octave icon_multimer_histogram.m "equal_levels_track_1*.mat"
#→ 得到 multimer_histogram.png, 就是你的 Fig. 4C-like 图.Figure Explanation (e.g. Track 14 and Track 100)
- Top panel (black line): Raw photon counts (
z).- Direct output from Step 1 HMM analysis; visualizes the original noisy fluorescence trace.
- Bottom panel:
- Gray line: Raw photon counts for direct comparison.
- Red line: Step-wise fit produced by discretizing the HMM mean (
m_mod) from Step 1 directly inside the python script. - Blue “▲”: Detected binding (upward) events.
- Black “▼”: Detected bleaching (downward) events.
- Event Table: Both “binding” and “bleach” events are exported with details: time, photon count, state transition, and dwell time.
Note:
- For these figures, only Step 1 and Step 3 are used.
- Step 2 is for diagnostic/discretization, but in our current pipeline,
L_bestwas given directly with 10, was not calculated from Step 2, therefore Step 2 was not used. - Step 4 is left for future population summaries.
Key Script Function Descriptions
1. icon_from_track_csv.m (Octave/MATLAB)
- Loads a csv photon count time sequence for a single molecule track.
- Infers hidden states and mean trajectory with ICON/HMM.
- Saves all variables for python to use.
2. plot_fig4AB_python.py (Python)
- Loads
.matresults: time (t), photons (z), and HMM means (m_mod). - Discretizes the mean trajectory into L equal steps, maps continuous states to nearest discrete, and fits photon counts by linear regression for step heights.
- Detects step transitions corresponding to binding or bleaching based on user parameters (size thresholds, dwell filters).
- Plots raw data + stepwise fit, annotates events, and saves tables.
(Script excerpt, see full file for details):
def assign_to_levels(m_mod, levels):
# Map every m_mod value to the nearest discrete level
...
def detect_binding_bleach_from_state(...):
# Identify up/down steps using given jump sizes and baseline cutoff
...
def filter_short_episodes_by_dwell(...):
# Filter events with insufficient dwell time
...
...
if __name__ == "__main__":
# Parse command line args, load .mat, process and plotComplete Scripts
See attached files for full script code:
icon_from_track_csv.mplot_fig4AB_python.pyicon_post_equal_levels.m(diagnostic, not used for current figures)icon_multimer_histogram.m(future)
Example Event Table Output
The python script automatically produces a CSV/Excel file summarizing:
- Event type (“binding” or “bleach”)
- Time (seconds)
- Photon count (at event)
- States before/after the event
- Dwell time (for binding)
In summary:
- Figures output from
plot_fig4AB_python.pydirectly visualize both binding and bleaching events as blue (▲) and black (▼) markers, using logic based on HMM analysis and transition detection within the Python code, without any direct dependence on Step 2 “equal_levels” files. This approach is both robust and reproducible for detailed single-molecule state analysis. [^1][^2]
icon_from_track_csv.m
clear all;
%% icon_from_track_csv.m
%% 用 ICON HMM 分析你自己的 track_intensity CSV 数据
%%
%% 用法(命令行):
%% octave icon_from_track_csv.m your_track_intensity_file.csv [track_id]
%%
%% - your_track_intensity_file.csv:
%% 3 列分号分隔:
%% # track index;time (seconds);track intensity (photon counts)
%% - track_id(可选):
%% 要分析的 track_index 数值,例如 0、2、10、100 等
%% 如果不给,则自动:
%% - 若存在 track 100,则用 100
%% - 否则用第一个 track
%%--------------------------------------------
%% 0. 处理命令行参数
%%--------------------------------------------
arg_list = argv();
if numel(arg_list) < 1
error("Usage: octave icon_from_track_csv.m
<track_intensity_csv> [track_id]");
end
input_file = arg_list{1};
track_id_arg = NaN;
if numel(arg_list) >= 2
track_id_arg = str2double(arg_list{2});
end
fprintf("Input CSV : %s\n", input_file);
if ~isnan(track_id_arg)
fprintf("Requested track_id: %g\n", track_id_arg);
end
%% 链接 ICON sampler 的源码
%addpath('sampler_SRC');
% 尝试加载 statistics 包(gamrnd 在这里)
try
pkg load statistics;
catch
warning("Could not load 'statistics' package. Please install it via 'pkg install -forge statistics'.");
end
%%--------------------------------------------
%% 1. 读入 3 列 CSV: track_index;time;counts
%%--------------------------------------------
fid = fopen(input_file, 'r');
if fid < 0
error("Cannot open file: %s", input_file);
end
% 第一行是注释头 "# track index;time (seconds);track intensity (photon counts)"
header_line = fgetl(fid); %#ok
<NASGU>
% 后面每行: track_index;time_sec;intensity
data = textscan(fid, "%f%f%f", "Delimiter", ";");
fclose(fid);
track_idx = data{1};
time_sec = data{2};
counts = data{3};
% 按 track 和时间排序,保证序列有序
[~, order] = sortrows([track_idx, time_sec], [1, 2]);
track_idx = track_idx(order);
time_sec = time_sec(order);
counts = counts(order);
tracks = unique(track_idx);
fprintf("Found %d tracks: ", numel(tracks));
fprintf("%g ", tracks);
fprintf("\n");
%%--------------------------------------------
%% 2. 选择要分析的 track
%%--------------------------------------------
if ~isnan(track_id_arg)
tr = track_id_arg;
else
% 如果存在 track 100,则优先选 100(你自己定义的 accumulated 轨迹)
if any(tracks == 100)
tr = 100;
else
tr = tracks(1); % 否则就选第一个
end
end
if ~any(tracks == tr)
error("Requested track_id = %g not found in file.", tr);
end
fprintf("Using track_id = %g for ICON analysis.\n", tr);
sel = (track_idx == tr);
t = time_sec(sel);
z = counts(sel);
% 按时间排序(理论上已经排过一次,这里再保险)
[ t, order_t ] = sort(t);
z = z(order_t);
z = z(:); % 列向量
N = numel(z);
fprintf("Track %g has %d time points.\n", tr, N);
%%--------------------------------------------
%% 3. 设置 ICON 参数(与原始脚本一致)
%%--------------------------------------------
% 浓度(Dirichlet 相关超参数)
opts.a = 1; % Transitions (alpha)
opts.g = 1; % Base (gamma)
% 超参数 Q
opts.Q(1) = mean(z); % mean of means (lambda)
opts.Q(2) = 1 / std(z)^2; % Precision of means (rho)
opts.Q(3) = 0.1; % Shape of precisions (beta)
opts.Q(4) = 0.00001; % Scale of precisions (omega)
opts.M = 10; % Nodes in the interpolation
% 采样器参数
opts.dr_sk = 1; % Stride: 每多少步保存一次 sample
opts.K_init = 50; % 初始 photon levels 数量(ICON 会自动收缩)
% 输出标志
flag_stat = true; % 在命令行里打印进度
flag_anim = false; % 是否弹出动画(Octave 下可以改成 false 更稳定)
%%--------------------------------------------
%% 4. 运行 ICON 采样器
%%--------------------------------------------
R = 1000; % 样本数,可按需要调整
fprintf("Running ICON sampler on track %g ...\n", tr);
chain = chainer_main( z, R, [], opts, flag_stat, flag_anim );
%%--------------------------------------------
%% 5. 导出 samples 方便后处理
%%--------------------------------------------
fr = 0.25; % burn-in 比例(前 25%% 样本丢掉)
dr = 2; % sample stride(每隔多少个样本导出一个)
out_prefix = sprintf('samples_track_%g', tr);
chainer_export(chain, fr, dr, out_prefix, 'mat');
fprintf("Samples exported to %s.mat\n", out_prefix);
%%--------------------------------------------
%% 6. 基本后验分析:state trajectory / transitions / drift
%%--------------------------------------------
% 离散化 state space (在 [0,1] 上划分 25 个 bin)
m_min = 0;
m_max = 1;
m_num = 25;
% (1) 状态轨迹(归一化):m_mod
[m_mod, m_red] = chainer_analyze_means(chain, fr, dr, m_min, m_max, m_num, z);
% (2) 转移概率
[m_edges, p_mean, d_dist] = chainer_analyze_transitions( ...
chain, fr, dr, m_min, m_max, m_num, true);
% (3) 漂移轨迹
[y_mean, y_std] = chainer_analyze_drift(chain, fr, dr, z);
% 1) Load the original Octave .mat file
%load('icon_analysis_track_100.mat');
% 2) Re-save in MATLAB-compatible v5/v7 format, with new name
%save('-mat', 'icon_analysis_track_100_matlab.mat');
%% 存到一个 mat 文件里,方便之后画图
%icon_mat = sprintf('icon_analysis_track_%g.mat', tr);
%save(icon_mat, 't', 'z', 'm_mod', 'm_red', 'm_edges', 'p_mean', ...
% 'd_dist', 'y_mean', 'y_std');
% 原来的 Octave 版本(可留可不留)
mat_out = sprintf("icon_analysis_track_%d.mat", tr);
save(mat_out, "m_mod", "m_red", "m_edges", "p_mean", "d_dist", ...
"y_mean", "y_std", "t", "z");
% 额外保存一个专门给 Python / SciPy 用的 MATLAB-compatible 版本
mat_out_matlab = sprintf("icon_analysis_track_%d_matlab.mat", tr);
save('-mat', mat_out_matlab, "m_mod", "m_red", "m_edges", "p_mean", "d_dist", ...
"y_mean", "y_std", "t", "z");
fprintf("ICON analysis saved to %s\n", icon_mat);
fprintf("Done.\n");icon_post_equal_levels.m
% icon_post_equal_levels.m (script version)
%
% 用法(终端):
% octave icon_post_equal_levels.m icon_analysis_track_XXX.mat
%
% 对 ICON 分析结果 (icon_analysis_track_XXX.mat) 做等间距 photon level 后处理
arg_list = argv();
if numel(arg_list) < 1
error("Usage: octave icon_post_equal_levels.m icon_analysis_track_XXX.mat");
end
mat_file = arg_list{1};
fprintf("Loading ICON analysis file: %s\n", mat_file);
S = load(mat_file);
if ~isfield(S, 'm_mod') || ~isfield(S, 't') || ~isfield(S, 'z')
error("File %s must contain variables: t, z, m_mod.", mat_file);
end
t = S.t(:);
z = S.z(:);
m_mod = S.m_mod(:);
N = numel(m_mod);
if numel(t) ~= N || numel(z) ~= N
error("t, z, m_mod must have the same length.");
end
% 从文件名里解析 track_id(如果有)
[~, name, ~] = fileparts(mat_file);
track_id = NaN;
tokens = regexp(name, 'icon_analysis_track_([0-9]+)', 'tokens');
if ~isempty(tokens)
track_id = str2double(tokens{1}{1});
fprintf("Detected track_id = %g from file name.\n", track_id);
else
fprintf("Could not parse track_id from file name, set to NaN.\n");
end
%--------------------------------------------
% 1. 在 m_mod 的范围上搜索等间距 level 数 L
%--------------------------------------------
m_min = min(m_mod);
m_max = max(m_mod);
fprintf("m_mod range: [%.4f, %.4f]\n", m_min, m_max);
L_min = 2;
L_max = 12; % 可以按需要改大一些
best_score = Inf;
L_best = L_min;
fprintf("Scanning candidate level numbers L = %d .. %d ...\n", L_min, L_max);
for L = L_min:L_max
levels = linspace(m_min, m_max, L);
% 把每个 m_mod(t) 映射到最近的 level:state_temp ∈ {1..L}
state_temp = assign_to_levels(m_mod, levels);
% 用这些 level 生成 step-wise 轨迹
m_step_temp = levels(state_temp);
% 计算拟合误差 SSE(在归一化空间)
residual = m_mod - m_step_temp;
sse = sum(residual.^2);
% 类 BIC 评分:误差 + 惩罚 L
score = N * log(sse / N + eps) + L * log(N);
fprintf(" L = %2d -> SSE = %.4g, score = %.4g\n", L, sse, score);
if score < best_score
best_score = score;
L_best = L;
end
end
fprintf("Best L (number of equally spaced levels) = %d\n", L_best);
%--------------------------------------------
% 2. 用最优 L_best 构建最终的等间距 level & state
%--------------------------------------------
levels_norm = linspace(m_min, m_max, L_best);
state = assign_to_levels(m_mod, levels_norm); % 1..L_best
m_step = levels_norm(state); % 归一化台阶轨迹
%--------------------------------------------
% 3. 将归一化台阶轨迹线性映射回 photon counts
% z ≈ a * m_step + b (最小二乘)
%--------------------------------------------
A = [m_step(:), ones(N,1)];
theta = A \ z; % 最小二乘拟合 [a; b]
a = theta(1);
b = theta(2);
z_step = A * theta; % 拟合出来的 photon counts 台阶轨迹
fprintf("Fitted z ≈ a * m_step + b with a = %.4f, b = %.4f\n", a, b);
%--------------------------------------------
% 4. 检测 bleaching 步:state 下降的时刻
%--------------------------------------------
%s_prev = state(1:end-1);
%s_next = state(2:end);
%
%bleach_idx = find(s_next < s_prev) + 1;
%bleach_times = t(bleach_idx);
%
%fprintf("Found %d bleaching step(s).\n", numel(bleach_idx));
%--------------------------------------------
% Detect upward (binding) and downward (bleach) steps
%--------------------------------------------
s_prev = state(1:end-1);
s_next = state(2:end);
% raw step indices
bind_idx_raw = find(s_next > s_prev) + 1; % upward jumps
bleach_idx_raw = find(s_next < s_prev) + 1; % downward jumps
% Optional: threshold by intensity change to ignore tiny noisy steps
dz = z_step(2:end) - z_step(1:end-1);
min_jump = 30; % <-- choose something ~ one level step or larger
keep_bind = dz > min_jump;
keep_bleach = dz < -min_jump;
bind_idx = bind_idx_raw(keep_bind(bind_idx_raw-1));
bleach_idx = bleach_idx_raw(keep_bleach(bleach_idx_raw-1));
bind_times = t(bind_idx);
bleach_times = t(bleach_idx);
fprintf("Found %d binding and %d bleaching steps (with threshold %.1f).\n", ...
numel(bind_idx), numel(bleach_idx), min_jump);
%--------------------------------------------
% 5. 保存结果
%--------------------------------------------
out_name = sprintf('equal_levels_track_%s.mat', ...
ternary(isnan(track_id), 'X', num2str(track_id)));
fprintf("Saving equal-level analysis to %s\n", out_name);
% Save them in the .mat file
%save(out_name, ...
% 't', 'z', 'm_mod', ...
% 'L_best', 'levels_norm', ...
% 'state', 'm_step', 'z_step', ...
% 'bleach_idx', 'bleach_times', ...
% 'track_id');
save(out_name, ...
't', 'z', 'm_mod', ...
'L_best', 'levels_norm', ...
'state', 'm_step', 'z_step', ...
'bind_idx', 'bind_times', ...
'bleach_idx', 'bleach_times', ...
'track_id');
fprintf("Done.\n");plot_fig4AB_python.py
#!/usr/bin/env python3
"""
plot_fig4AB_python.py
Usage:
python plot_fig4AB_python.py
<track_id> [L_best] [min_levels_bind] [min_levels_bleach] [dwell_min] [baseline_state_max]
Arguments
---------
<track_id> : int
e.g. 10 → uses icon_analysis_track_10_matlab.mat
[L_best] : int, optional
Number of equally spaced levels to use for the step-wise fit
in Fig. 4B. If omitted, default = 3.
[min_levels_bind] : int, optional
Minimum number of state levels for an upward jump to count as a
binding event. Default = 3.
[min_levels_bleach] : int, optional
Minimum number of state levels for a downward jump to count as a
bleaching step. Default = 1.
[dwell_min] : float, optional
Minimum allowed dwell time between a binding event and the NEXT
bleaching event. If Δt = t_bleach - t_bind < dwell_min, then:
- that binding is removed
- the paired bleaching event is also removed
Default = 0 (no dwell-based filtering).
[baseline_state_max] : int, optional
Highest state index (0-based) that is still considered "baseline /
unbound" before a binding jump.
Binding condition becomes:
dstate >= min_levels_bind AND state_before <= baseline_state_max
If omitted → no baseline constraint (any state can be a start).
Input file
----------
icon_analysis_track_
<track_id>_matlab.mat
Expected variables inside the .mat file:
t : time vector (1D, seconds)
z : photon counts (1D)
m_mod : ICON mean trajectory (1D), same length as t and z
Outputs
-------
1) Figure:
fig4AB_track_
<track_id>_L<L_best>.png
2) Event tables:
binding_bleach_events_track_
<track_id>_L<L_best>.csv
binding_bleach_events_track_
<track_id>_L<L_best>.xlsx (if pandas available)
"""
import sys
import os
import numpy as np
import matplotlib.pyplot as plt
from scipy.io import loadmat
# Try to import pandas for Excel output
try:
import pandas as pd
HAS_PANDAS = True
except ImportError:
HAS_PANDAS = False
print("[WARN] pandas not available: Excel output (.xlsx) will be skipped.")
def assign_to_levels(m_mod, levels):
"""
Map each point in m_mod to the nearest level.
Parameters
----------
m_mod : array-like, shape (N,)
Normalized ICON mean trajectory.
levels : array-like, shape (L,)
Candidate level values.
Returns
-------
state : ndarray, shape (N,)
Integer state indices in {0, 1, ..., L-1}.
"""
m_mod = np.asarray(m_mod).ravel()
levels = np.asarray(levels).ravel()
diff = np.abs(m_mod[:, None] - levels[None, :]) # (N, L)
state = np.argmin(diff, axis=1) # 0..L-1
return state
def detect_binding_bleach_from_state(
z_step,
t,
state,
levels,
min_levels_bind=3,
min_levels_bleach=1,
baseline_state_max=None,
):
"""
Detect binding (big upward state jumps) and bleaching (downward jumps)
using the discrete state sequence.
- Binding: large upward jump (>= min_levels_bind) starting from a
"baseline" state (state_before <= baseline_state_max) if that
parameter is given.
- Bleaching: downward jump (<= -min_levels_bleach)
Parameters
----------
z_step : array-like, shape (N,)
Step-wise photon counts.
t : array-like, shape (N,)
Time vector (seconds).
state : array-like, shape (N,)
Integer states 0..L-1 from assign_to_levels().
levels : array-like, shape (L,)
Level values (in m_mod space, not directly photon counts).
min_levels_bind : int
Minimum number of levels for an upward jump to be
considered a binding event.
min_levels_bleach : int
Minimum number of levels for a downward jump to be
considered a bleaching event.
baseline_state_max : int or None
Highest state index considered "baseline" before binding.
If None, any state can be the start of a binding jump.
Returns
-------
bind_idx, bleach_idx : np.ndarray of indices
bind_times, bleach_times : np.ndarray of times (seconds)
bind_values, bleach_values : np.ndarray of photon counts at those events
bind_state_before, bind_state_after : np.ndarray of integer states
bleach_state_before, bleach_state_after : np.ndarray of integer states
"""
z_step = np.asarray(z_step).ravel()
t = np.asarray(t).ravel()
state = np.asarray(state).ravel()
levels = np.asarray(levels).ravel()
N = len(t)
dstate = np.diff(state) # length N-1
idx = np.arange(N - 1)
# ----- Binding: big upward jump, optionally from baseline only -----
bind_mask = (dstate >= min_levels_bind)
if baseline_state_max is not None:
bind_mask &= (state[idx] <= baseline_state_max)
bind_idx = idx[bind_mask] + 1
# ----- Bleaching: downward jump -----
bleach_mask = (dstate <= -min_levels_bleach)
bleach_idx = idx[bleach_mask] + 1
bind_times = t[bind_idx]
bleach_times = t[bleach_idx]
bind_values = z_step[bind_idx]
bleach_values = z_step[bleach_idx]
bind_state_before = state[bind_idx - 1]
bind_state_after = state[bind_idx]
bleach_state_before = state[bleach_idx - 1]
bleach_state_after = state[bleach_idx]
return (
bind_idx,
bleach_idx,
bind_times,
bleach_times,
bind_values,
bleach_values,
bind_state_before,
bind_state_after,
bleach_state_before,
bleach_state_after,
)
def filter_short_episodes_by_dwell(
bind_idx,
bleach_idx,
bind_times,
bleach_times,
bind_values,
bleach_values,
bind_state_before,
bind_state_after,
bleach_state_before,
bleach_state_after,
dwell_min,
):
"""
Remove short binding episodes based on dwell time and also remove
the paired bleaching step.
Rule:
For each binding, find the first bleaching with t_bleach > t_bind.
If Δt = t_bleach - t_bind < dwell_min, then:
- remove this binding
- remove this bleaching
All other bleaching events remain.
Returns
-------
(filtered binding arrays, dwell_times) + filtered bleaching arrays
"""
if dwell_min <= 0 or len(bind_idx) == 0 or len(bleach_idx) == 0:
# no filtering requested or missing events
dwell_times = np.full(len(bind_idx), np.nan)
for i in range(len(bind_idx)):
future = np.where(bleach_times > bind_times[i])[0]
if len(future) > 0:
dwell_times[i] = bleach_times[future[0]] - bind_times[i]
return (
bind_idx,
bleach_idx,
bind_times,
bleach_times,
bind_values,
bleach_values,
bind_state_before,
bind_state_after,
bleach_state_before,
bleach_state_after,
dwell_times,
)
bind_idx = np.asarray(bind_idx)
bleach_idx = np.asarray(bleach_idx)
bind_times = np.asarray(bind_times)
bleach_times = np.asarray(bleach_times)
bind_values = np.asarray(bind_values)
bleach_values = np.asarray(bleach_values)
bind_state_before = np.asarray(bind_state_before)
bind_state_after = np.asarray(bind_state_after)
bleach_state_before = np.asarray(bleach_state_before)
bleach_state_after = np.asarray(bleach_state_after)
keep_bind = np.ones(len(bind_idx), dtype=bool)
keep_bleach = np.ones(len(bleach_idx), dtype=bool)
dwell_times = np.full(len(bind_idx), np.nan)
removed_bind = 0
removed_bleach = 0
for i in range(len(bind_idx)):
t_b = bind_times[i]
future = np.where(bleach_times > t_b)[0]
if len(future) == 0:
# no bleaching afterwards → dwell undefined, keep binding
dwell_times[i] = np.nan
continue
j = future[0]
dt = bleach_times[j] - t_b
dwell_times[i] = dt
if dt < dwell_min:
# remove this binding and its paired bleaching
keep_bind[i] = False
if keep_bleach[j]:
keep_bleach[j] = False
removed_bleach += 1
removed_bind += 1
print(
f"[INFO] Dwell-based filter: removed {removed_bind} binding(s) and "
f"{removed_bleach} paired bleaching step(s) with Δt < {dwell_min} s; "
f"{np.sum(keep_bind)} binding(s) and {np.sum(keep_bleach)} bleaching step(s) kept."
)
# Apply masks
bind_idx = bind_idx[keep_bind]
bind_times = bind_times[keep_bind]
bind_values = bind_values[keep_bind]
bind_state_before = bind_state_before[keep_bind]
bind_state_after = bind_state_after[keep_bind]
dwell_times = dwell_times[keep_bind]
bleach_idx = bleach_idx[keep_bleach]
bleach_times = bleach_times[keep_bleach]
bleach_values = bleach_values[keep_bleach]
bleach_state_before = bleach_state_before[keep_bleach]
bleach_state_after = bleach_state_after[keep_bleach]
return (
bind_idx,
bleach_idx,
bind_times,
bleach_times,
bind_values,
bleach_values,
bind_state_before,
bind_state_after,
bleach_state_before,
bleach_state_after,
dwell_times,
)
def plot_fig4AB(
track_id,
L_best=None,
min_levels_bind=3,
min_levels_bleach=1,
dwell_min=0.0,
baseline_state_max=None,
):
# --------------------------
# 1. Load ICON analysis file
# --------------------------
mat_file = f"icon_analysis_track_{track_id}_matlab.mat"
print(f"Loading {mat_file}")
if not os.path.exists(mat_file):
raise FileNotFoundError(f"{mat_file} does not exist in current directory.")
data = loadmat(mat_file)
def extract_vector(name):
if name not in data:
raise KeyError(f"Variable '{name}' not found in {mat_file}")
v = data[name]
return np.squeeze(v)
t = extract_vector("t")
z = extract_vector("z")
m_mod = extract_vector("m_mod")
if not (len(t) == len(z) == len(m_mod)):
raise ValueError("t, z, and m_mod must have the same length.")
N = len(t)
print(f"Track {track_id}: N = {N} points")
# --------------------------
# 2. Choose L (number of levels)
# --------------------------
if L_best is None:
L_best = 3 # fallback default
print(f"No L_best provided, using default L_best = {L_best}")
else:
print(f"Using user-specified L_best = {L_best}")
m_min = np.min(m_mod)
m_max = np.max(m_mod)
levels = np.linspace(m_min, m_max, L_best)
print(f"m_mod range: [{m_min:.4f}, {m_max:.4f}]")
print(f"Equally spaced levels ({L_best}): {levels}")
# --------------------------
# 3. Build step-wise trajectory from m_mod
# --------------------------
state = assign_to_levels(m_mod, levels) # 0..L_best-1
m_step = levels[state]
# Map back to photon counts via linear fit z ≈ a*m_step + b
A = np.column_stack([m_step, np.ones(N)])
theta, *_ = np.linalg.lstsq(A, z, rcond=None)
a, b = theta
z_step = A @ theta
print(f"Fitted z ≈ a * m_step + b with a = {a:.4f}, b = {b:.4f}")
# --------------------------
# 4. Detect binding / bleaching events (state-based)
# --------------------------
(
bind_idx,
bleach_idx,
bind_times,
bleach_times,
bind_values,
bleach_values,
bind_state_before,
bind_state_after,
bleach_state_before,
bleach_state_after,
) = detect_binding_bleach_from_state(
z_step,
t,
state,
levels,
min_levels_bind=min_levels_bind,
min_levels_bleach=min_levels_bleach,
baseline_state_max=baseline_state_max,
)
base_msg = (
f"baseline_state_max={baseline_state_max}"
if baseline_state_max is not None
else "baseline_state_max=None (no baseline restriction)"
)
print(
f"Initial detection: {len(bind_idx)} binding and {len(bleach_idx)} bleaching "
f"events (min_levels_bind={min_levels_bind}, "
f"min_levels_bleach={min_levels_bleach}, {base_msg})."
)
# --------------------------
# 5. Apply dwell-time filter to binding + paired bleach
# --------------------------
(
bind_idx,
bleach_idx,
bind_times,
bleach_times,
bind_values,
bleach_values,
bind_state_before,
bind_state_after,
bleach_state_before,
bleach_state_after,
dwell_times,
) = filter_short_episodes_by_dwell(
bind_idx,
bleach_idx,
bind_times,
bleach_times,
bind_values,
bleach_values,
bind_state_before,
bind_state_after,
bleach_state_before,
bleach_state_after,
dwell_min=dwell_min,
)
print(
f"After dwell filter (dwell_min={dwell_min}s): "
f"{len(bind_idx)} binding and {len(bleach_idx)} bleaching events remain."
)
# --------------------------
# 6. Build event table & save CSV / Excel
# --------------------------
rows = []
# Binding events
for i in range(len(bind_idx)):
idx = int(bind_idx[i])
rows.append({
"event_type": "binding",
"sample_index": idx,
"time_seconds": float(bind_times[i]),
"photon_count": float(bind_values[i]),
"state_before": int(bind_state_before[i]),
"state_after": int(bind_state_after[i]),
"level_before_norm": float(levels[bind_state_before[i]]),
"level_after_norm": float(levels[bind_state_after[i]]),
"dwell_time": float(dwell_times[i]) if not np.isnan(dwell_times[i]) else "",
})
# Bleaching events
for i in range(len(bleach_idx)):
idx = int(bleach_idx[i])
rows.append({
"event_type": "bleach",
"sample_index": idx,
"time_seconds": float(bleach_times[i]),
"photon_count": float(bleach_values[i]),
"state_before": int(bleach_state_before[i]),
"state_after": int(bleach_state_after[i]),
"level_before_norm": float(levels[bleach_state_before[i]]),
"level_after_norm": float(levels[bleach_state_after[i]]),
"dwell_time": "",
})
# Sort by time
rows = sorted(rows, key=lambda r: r["time_seconds"])
# Write CSV
csv_name = f"binding_bleach_events_track_{track_id}_L{L_best}.csv"
import csv
with open(csv_name, "w", newline="") as f:
writer = csv.DictWriter(
f,
fieldnames=[
"event_type",
"sample_index",
"time_seconds",
"photon_count",
"state_before",
"state_after",
"level_before_norm",
"level_after_norm",
"dwell_time",
],
)
writer.writeheader()
for r in rows:
writer.writerow(r)
print(f"Saved event table to {csv_name}")
# Write Excel (if pandas available)
if HAS_PANDAS:
df = pd.DataFrame(rows)
xlsx_name = f"binding_bleach_events_track_{track_id}_L{L_best}.xlsx"
df.to_excel(xlsx_name, index=False)
print(f"Saved event table to {xlsx_name}")
else:
print("[INFO] pandas not installed → skipped Excel (.xlsx) output.")
# --------------------------
# 7. Make a figure similar to Fig. 4A + 4B
# --------------------------
fig, axes = plt.subplots(2, 1, figsize=(7, 6), sharex=True)
# ---- Fig. 4A-like: raw intensity vs time ----
ax1 = axes[0]
ax1.plot(t, z, color="black", linewidth=0.8)
ax1.set_ylabel("Photon counts")
ax1.set_title(f"Track {track_id}: raw intensity") #(Fig. 4A-like)
# ---- Fig. 4B-like: step-wise HMM fit vs time ----
ax2 = axes[1]
ax2.plot(t, z, color="0.8", linewidth=0.5, label="raw")
ax2.plot(t, z_step, color="red", linewidth=1.5,
label=f"equal levels (L={L_best})")
# Mark binding (up-steps) and bleaching (down-steps) AFTER filtering
if len(bind_idx) > 0:
ax2.scatter(
bind_times,
bind_values,
marker="^",
color="blue",
s=30,
label="binding",
)
if len(bleach_idx) > 0:
ax2.scatter(
bleach_times,
bleach_values,
marker="v",
color="black",
s=30,
label="bleach",
)
ax2.set_xlabel("Time (s)")
ax2.set_ylabel("Photon counts")
ax2.set_title(
f"Step-wise HMM fit (" #Fig. 4B-like,
f"min_bind_levels={min_levels_bind}, "
f"min_bleach_levels={min_levels_bleach}, "
f"dwell_min={dwell_min}s, "
f"baseline_state_max={baseline_state_max})"
)
ax2.legend(loc="best")
fig.tight_layout()
out_png = f"fig4AB_track_{track_id}_L{L_best}.png"
fig.savefig(out_png, dpi=300)
plt.close(fig)
print(f"Saved figure to {out_png}")
if __name__ == "__main__":
if len(sys.argv) < 2:
print("Usage: python plot_fig4AB_python.py
<track_id> [L_best] [min_levels_bind] [min_levels_bleach] [dwell_min] [baseline_state_max]")
sys.exit(1)
track_id = int(sys.argv[1])
# Defaults
L_best = None
min_levels_bind = 3
min_levels_bleach = 1
dwell_min = 0.0
baseline_state_max = None
if len(sys.argv) >= 3:
L_best = int(sys.argv[2])
if len(sys.argv) >= 4:
min_levels_bind = int(sys.argv[3])
if len(sys.argv) >= 5:
min_levels_bleach = int(sys.argv[4])
if len(sys.argv) >= 6:
dwell_min = float(sys.argv[5])
if len(sys.argv) >= 7:
baseline_state_max = int(sys.argv[6])
plot_fig4AB(
track_id,
L_best=L_best,
min_levels_bind=min_levels_bind,
min_levels_bleach=min_levels_bleach,
dwell_min=dwell_min,
baseline_state_max=baseline_state_max,
)icon_multimer_histogram.m
% icon_multimer_histogram.m
%
% 用法(终端):
% octave icon_multimer_histogram.m [pattern]
%
% 默认 pattern = "equal_levels_track_*.mat"
%
% 要求每个 mat 文件里至少有:
% state : 等间距 level 的索引 (1..L_best)
% t : 时间向量
% z : 原始 photon counts
% track_id : (可选) 轨迹编号,用于打印信息
%
% 输出:
% - 在命令行打印每条轨迹估计出的 multimer 数目
% - 生成 Fig. 4C 风格直方图:
% multimer_histogram.png
arg_list = argv();
if numel(arg_list) >= 1
pattern = arg_list{1};
else
pattern = "equal_levels_track_*.mat";
end
fprintf("Searching files with pattern: %s\n", pattern);
files = dir(pattern);
if isempty(files)
error("No files matched pattern: %s", pattern);
end
multimers = []; % 保存每条轨迹的 multimer size
track_ids = []; % 保存对应的 track_id(若存在)
fprintf("Found %d files.\n", numel(files));
for i = 1:numel(files)
fname = files(i).name;
fprintf("\nLoading %s ...\n", fname);
S = load(fname);
if ~isfield(S, "state") || ~isfield(S, "t")
warning(" File %s does not contain 'state' or 't'. Skipped.", fname);
continue;
end
state = S.state(:);
t = S.t(:);
N = numel(state);
if N < 5
warning(" File %s has too few points (N=%d). Skipped.", fname, N);
continue;
end
% 解析 track_id(如果有)
tr_id = NaN;
if isfield(S, "track_id")
tr_id = S.track_id;
else
% 尝试从文件名里解析
tokens = regexp(fname, 'equal_levels_track_([0-9]+)', 'tokens');
if ~isempty(tokens)
tr_id = str2double(tokens{1}{1});
end
end
% 取前 10%% 和后 10%% 时间段的 state 中位数
n_head = max(1, round(0.1 * N));
n_tail = max(1, round(0.1 * N));
head_idx = 1:n_head;
tail_idx = (N - n_tail + 1):N;
initial_state = round(median(state(head_idx)));
final_state = round(median(state(tail_idx)));
multimer_size = initial_state - final_state;
if multimer_size <= 0
fprintf(" Track %g: initial_state=%d, final_state=%d -> multimer_size=%d (ignored)\n", ...
tr_id, initial_state, final_state, multimer_size);
continue;
end
fprintf(" Track %g: initial_state=%d, final_state=%d -> multimer_size=%d\n", ...
tr_id, initial_state, final_state, multimer_size);
multimers(end+1,1) = multimer_size;
track_ids(end+1,1) = tr_id;
end
if isempty(multimers)
error("No valid multimer sizes estimated. Check your data or thresholds.");
end
% 像论文那样,可以选择去掉 monomer/dimer
fprintf("\nTotal %d events (including monomer/dimer).\n", numel(multimers));
% 可选:过滤掉 ≤2 的(monomer / dimer)
mask = multimers > 2;
multimers_filtered = multimers(mask);
fprintf("After removing monomer/dimer (<=2): %d events.\n", numel(multimers_filtered));
if isempty(multimers_filtered)
error("No events left after filtering monomer/dimer. Try including them.");
end
% 计算直方图
max_mult = max(multimers_filtered);
edges = 0.5:(max_mult + 0.5);
[counts, edges_out] = histcounts(multimers_filtered, edges);
centers = 1:max_mult;
% 画 Fig. 4C 风格的柱状图
figure;
bar(centers, counts, 'FaceColor', [0.2 0.6 0.8]); % 颜色随便,Octave 会忽略
xlabel('Number of mN-LT per origin (multimer size)');
ylabel('Frequency');
title('Distribution of multimer sizes (Fig. 4C-like)');
xlim([0.5, max_mult + 0.5]);
% 在每个柱子上标一下计数
hold on;
for k = 1:max_mult
if counts(k) > 0
text(centers(k), counts(k) + 0.1, sprintf('%d', counts(k)), ...
'HorizontalAlignment', 'center');
end
end
hold off;
print('-dpng', 'multimer_histogram.png');
fprintf("\n[INFO] Multimer histogram saved to multimer_histogram.png\n");Bioinformatics Pipelines for DNA Sequencing: From Raw Reads to Biological Insight
Abstract
English: Advances in DNA sequencing have revolutionized biology, but converting vast sequencing data into usable, robust biological knowledge depends on sophisticated bioinformatics. This review details computational strategies spanning all phases of DNA sequence analysis, starting from raw reads through to functional interpretation and reporting. It begins by characterizing the main sequencing platforms (short-read, long-read, targeted, and metagenomic), describes critical pipeline steps (sample tracking, quality control, read alignment, error correction, variant and structural variant detection, copy number analysis, de novo assembly), and considers the impact of reference genome choice and computational algorithms. Recent machine learning advances for variant annotation and integration with other omics are discussed, with applications highlighted in rare disease diagnostics, cancer genomics, and infectious disease surveillance. Emphasis is placed on reproducible, scalable, and well-documented pipelines using open-source tools, workflow management (Snakemake, Nextflow), containerization, versioning, and FAIR data principles. The review concludes with discussion of ongoing challenges (heterogeneous data, batch effects, benchmarking, privacy) and practical recommendations for robust, interpretable analyses for both experimental biologists and computational practitioners.
Chinese: DNA测序的持续进步彻底改变了生物学和医学研究,而要将庞大的测序数据转化为可靠的生物学知识,则高度依赖高水平的生物信息学方法。本文详细介绍了DNA序列分析全流程的主流计算策略,涵盖原始reads到功能性注释乃至标准化报告的各个环节。首先评述主流测序技术平台(短读长读、靶向、宏基因组),系统阐述实验设计、样本追踪、数据质控、比对、纠错、变异与结构变异检测、拷贝数分析和de novo组装等流程要点,并分析参考基因组和比对算法对结果的影响。文章还总结了机器学习在变异注释、多组学整合中的最新进展,结合罕见病诊断、肿瘤基因组和病原体监测等实际案例深入说明其应用场景。着重强调可重复、高效、透明的分析流程,包括开源工具、流程管理(Snakemake、Nextflow)、容器化、版本管理与FAIR原则。最后讨论了异质数据、批次效应、评测标准和隐私保护等挑战,并为实验与计算生物学研究者提供实用建议。
Detailed Structure \& Outline
- Introduction
- Historical overview of DNA sequencing and bioinformatics development
- The necessity of bioinformatics for handling scale, complexity, and error sources in modern sequence data
- Scope: DNA focus (excluding RNA/proteome)
- DNA Sequencing Technologies \& Data Properties
- 2.1 Short-read platforms (e.g., Illumina): read length, quality, use cases
- 2.2 Long-read platforms (PacBio, Nanopore): strengths, error profiles, applications
- 2.3 Specialized applications: targeted/exome panels, metagenomics, amplicon/barcode-based diagnostics
- Core Bioinformatics Pipeline Components
- 3.1 Experimental metadata, sample barcoding, batch tracking: Crucial for reproducibility and QC
- 3.2 Raw read QC: base quality, adapter/contaminant trimming, typical software/plots
- 3.3 Read alignment/mapping: reference choice (GRCh38, hg19), algorithmic details (FM-index, seed-and-extend), uniqueness/multimapping
- 3.4 Post-alignment processing: file sorting, duplicate marking, base recalibration, depth analysis
- 3.5 Variant calling: SNVs/indels, somatic vs germline separation, quality filters and validation strategies
- 3.6 Structural variant and CNV analysis: breakpoints, split/discordant reads, long-read tools
- 3.7 De novo assembly, polishing, and consensus generation where relevant
- Functional Interpretation
- 4.1 Annotation: gene models, regulatory regions, predictive algorithms and public databases
- 4.2 Multi-omics integration: joint analysis of genome, epigenome, transcriptome, regulatory networks
- 4.3 Machine learning/AI approaches: variant scoring, prioritization, deep learning for sequence features
- Reproducible and Scalable Workflows
- 5.1 Pipeline frameworks: Snakemake, Nextflow, CWL, and workflow description languages
- 5.2 Containerization: Docker, Singularity for reproducible deployments
- 5.3 Version control/documentation: workflow hubs, deployment on GitHub, FAIR-compliant reporting
- 5.4 Data management: standard formats (FASTQ/BAM/CRAM/VCF), secure storage, metadata
- Applications \& Case Studies
- Rare disease genomics: WGS for diagnosis
- Cancer genomics: tumor heterogeneity, evolution, therapy response
- Pathogen surveillance: rapid outbreak detection, resistance tracking
- Other applications to match research interests
- Challenges and Future Prospects
- Technical: population-scale analysis, batch correction, pangenomes, benchmarking complexities
- Practical: workflow sharing, legal/ethical/privacy issues
- Methodological: handling new sequencing chemistries, multi-modal omics
- Conclusions
- Recap essential lessons
- Actionable recommendations for robust design and execution
- Prospects for further automation, integration, and clinical translation
Section Opening (English / 中文): High-throughput DNA sequencing has fundamentally transformed modern genomics, enabling detailed investigation of human diseases, microbial ecology, and evolution. However, the raw output—massive quantities of short or long reads—is only the starting point; extracting meaningful, robust insights requires optimized bioinformatics pipelines that ensure data integrity and biological relevance.
高通量DNA测序极大地推动了现代基因组学,助力人类疾病、微生物生态与进化等领域的深入探索。但测序仪输出的原始reads只是起点——要获得有意义、可靠的生物学结论,必须依赖优化的生物信息学流程以保证数据质量和生物学解释的可信度。